
Erdmann Group
Compartimenti separati in fase liquida e necessità di targeting subcellulare
La compartimentazione è una caratteristica dell’organizzazione cellulare. Serve a separare reazioni chimiche incompatibili, consente la creazione di microambienti specializzati e può rendere più efficienti le reazioni chimiche limitando a volumi parziali gli attori molecolari coinvolti. Tradizionalmente, si riferisce a compartimenti legati alla membrana quali il reticolo endoplasmatico (ER), l’apparato di Golgi o i mitocondri. Le membrane in questi organelli non solo rappresentano interfacce di separazione, ma possono anche fungere da piattaforme bidimensionali per l’organizzazione dei percorsi di reazione.
Negli ultimi anni è emerso un nuovo concetto di organizzazione citoplasmatica: compartimenti privi di membrana risultanti dalla separazione di fase liquido-liquido (LLPS). Esempi di tali compartimenti negli eucarioti includono i corpi di Cajal e il nucleolo. È sempre più evidente che la LLPS è di grande importanza per comprendere la patogenesi di diverse malattie trasmissibili, disturbi neurodegenerativi e invecchiamento.
Di conseguenza, sono necessari metodi che consentano di studiare la separazione di fase liquido-liquido all’interno di cellule intatte (“in situ”).
Per rispondere a questa esigenza, utilizziamo una combinazione di microscopia a crio-fluorescenza (FLM), crio-lift out (LO), fresatura a fascio ionico crio-focalizzato (FIB) e tomografia crioelettronica (ET) per studiare la fase liquido-liquido in compartimenti separati all’interno di cellule e tessuti vetrificati. Ci sforziamo costantemente di migliorare l’accuratezza di questa pipeline sviluppando nuovi software e hardware.
Studi recenti e in corso nel nostro gruppo:
Utilizzando l’approccio FIB correlativo 3D (Fig. 1), stiamo studiando l’ultrastruttura sia dei compartimenti naturali, sia di compartimenti separati fase liquido-liquido formati durante i processi patologici. Recentemente, abbiamo fornito approfondimenti sull’organizzazione del nucleolo, un esempio di LLPS costitutivo in tutte le cellule eucariotiche e luogo della biogenesi dei ribosomi. Lavorando su alghe, lieviti e cellule di mammiferi, possiamo seguire la formazione della subunità ribosomale piccola e grande su un’ampia gamma dell’albero evolutivo, rivelando nuovi intermedi che potrebbero essere stati persi da esperimenti con particelle singole (“single particle”).
Oltre al nucleolo, ci sono numerosi compartimenti LLPS transitori all’interno delle cellule per i quali manca ancora una descrizione ultrastrutturale. In una collaborazione con Florian Wilfling (MPI for Biophysics, Francoforte), abbiamo recentemente scoperto e descritto l’ultrastruttura di un nuovo compartimento separato in fase liquida coinvolto nella cancellazione di eventi endocitici falliti in S. cerevisiae. Questo LLPS, che abbiamo chiamato “END” per il deposito di proteine endocitiche, aiuta a tamponare le proteine dell’endocitosi precoce e le cede al meccanismo autofagico, se necessario.
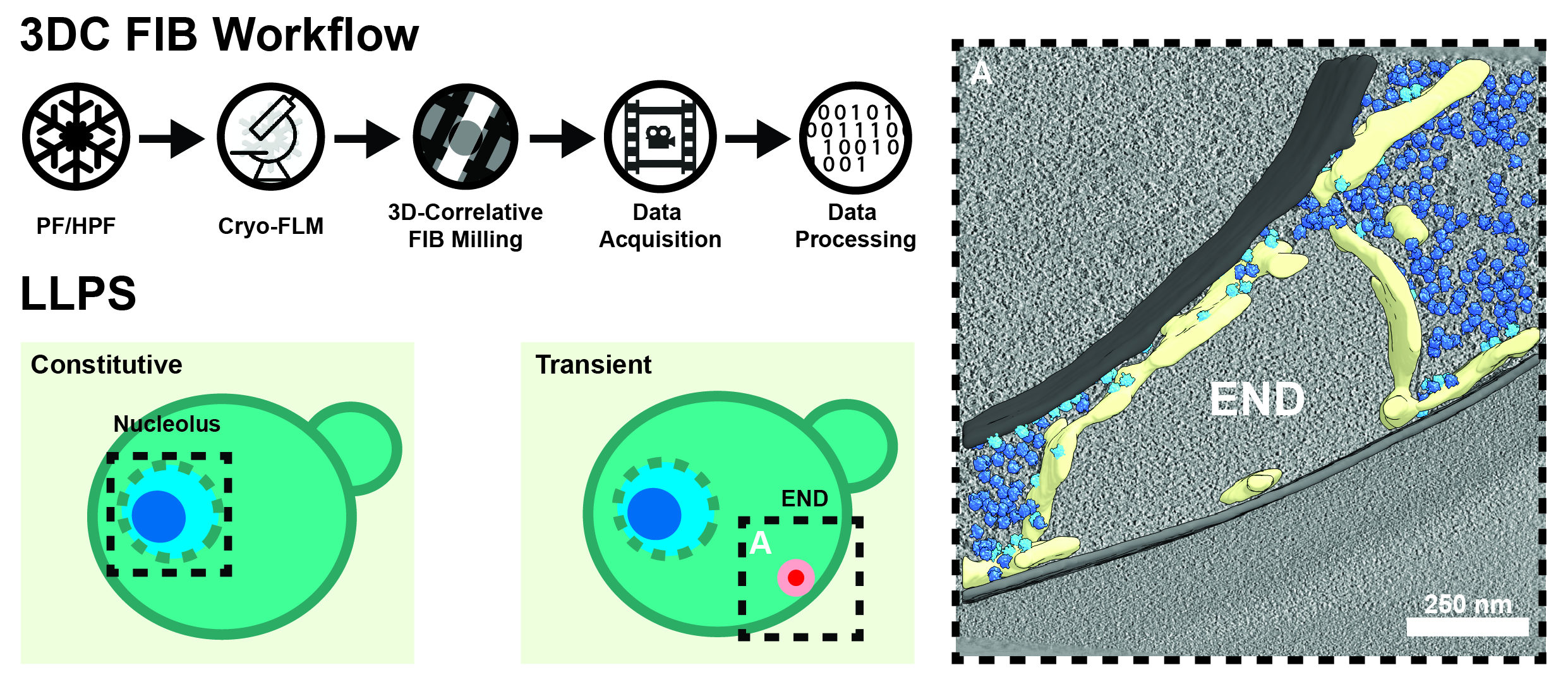
Figura 1. Fresatura 3D-Correlative (3DC) con fascio ionico focalizzato (FIB). Partendo da campioni congelati a immersione (PF) o congelati ad alta pressione (HPF), eseguiamo microscopia ottica confocale criogenica a fluorescenza (FLM) per localizzare le caratteristiche di interesse. Utilizzando queste informazioni, miriamo alle nostre lamelle durante la fresatura FIB prima che siano sottoposte all’acquisizione e all’elaborazione automatizzata del crio-tomogramma. Abbiamo già utilizzato questa tecnica su una varietà di compartimenti LLP, tra cui il nucleolo dell’alga verde C. reinhardtii e il deposito di proteine endocitiche (END), che gioca un ruolo critico nell’omeostasi delle prime proteine endocitiche in S. cerevisiae (a destra; membrane = grigio, ER = giallo, ribosomi liberi = blu scuro, ribosomi associati a ER = azzurro).
La localizzazione e il targeting di strutture subcellulari come i compartimenti separati dalla fase liquido-liquido, tuttavia, è solo l’inizio della storia. Comprendere la loro struttura interna potrebbe potenzialmente aiutare a chiarire il loro ruolo nella normale funzione cellulare e nella malattia. Quando si lavora in ambienti cellulari complessi come i campioni di tessuto, tuttavia, stabilire e confermare l’identità delle cellule osservate da TEM può essere difficile, nonostante la tecnica crio-FLM sia a nostra disposizione. Ad esempio, come distinguere tra intestino, nervo e cellule riproduttive in campioni di C. elegans congelati ad alta pressione a livello TEM (Fig. 2, a sinistra)?
C’è chiaramente bisogno sia di cryo-markers che di tag che possano essere usati per stabilire a) l’identità cellulare e b) localizzare le proteine, che sono troppo piccole o troppo disordinate per essere riconosciute direttamente da cryo-TEM. Per determinare l’identità cellulare, utilizziamo complessi proteici naturali (encapsuline batteriche, EncA) e complessi proteici progettati ex novo, che possono essere espressi sotto promotori tessuto-specifici. Questi mega-complessi Dalton possono essere facilmente rilevati anche in ambienti molto affollati come le sinapsi ippocampali di topo (Fig. 2, al centro). Tuttavia, gli EncAs non possono essere utilizzati come contrassegno per singole proteine a causa della loro dimensione e molteplicità di legame. Pertanto, stiamo sviluppando contrassegni (tags) basati su piccole proteine artificiali, che possono essere direttamente collegate ai bersagli di interesse e che quindi possono trasmettere la loro posizione per mezzo di una forma caratteristica. A differenza dei marcatori EncA, per questo sono necessari design proteici molto più piccoli, ma allo stesso tempo ancora rilevabili e dalla forma unica. Un compito impegnativo per il nostro team e i nostri collaboratori!
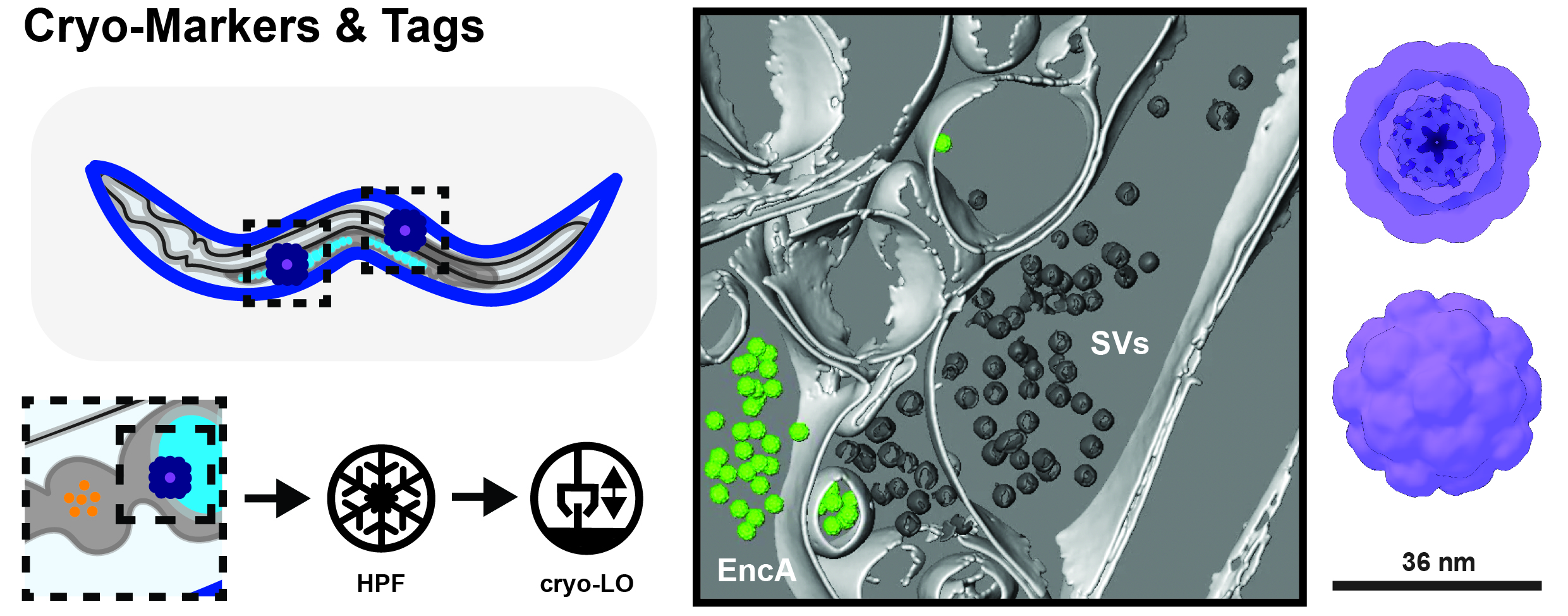
Figura 2. Crio marcatori e contrassegni (Tags) per il lavoro in situ. Stabilire l’identità cellulare nei campioni di crio-lift out (LO) può essere difficile poiché non ci sono molte caratteristiche endogene da cui partire (a sinistra). Quindi, stiamo usando complessi proteici non reattivi come le encapsuline (EncAs), che possono essere espresse sotto promotori tessuto-specifici. Questi possono essere facilmente rilevati ad es. nelle sinapsi dei neuroni dissociati dell’ippocampo di topo (al centro nella figura 2 – membrane = grigio chiaro; vescicole sinaptiche = grigio scuro; EncA = verde).
Membri del gruppo
-
 Laura Bonanomi
Laura Bonanomi
PhD Student -
 Philipp Sebastian Erdmann
Philipp Sebastian Erdmann
Research Group Leader -
 Jasmine Nguyen
Jasmine Nguyen
PhD Student -
 Gaia Perone
Gaia Perone
PhD Student -
 Jessica Winter
Jessica Winter
Scientific Visitor
