Il machine learning rivela caratteristiche nascoste dei tessuti umani
I ricercatori di Human Technopole hanno sviluppato un modello di machine learning auto-supervisionato che combina istologia, espressione genica e variazione genetica per identificare e raggruppare automaticamente distinte sottostrutture tissutali, cellule e tratti patologici nei tessuti umani.
L’istologia è una tecnica che consente l’identificazione microscopica di diversi componenti e strutture cellulari in un tessuto. L’esame istologico dei tessuti è fondamentale per diagnosticare accuratamente le malattie e fornisce informazioni cruciali nelle diagnosi cliniche. Tradizionalmente, i patologi esaminano sezioni tissutali colorate al microscopio. Tuttavia, l’avvento della digitalizzazione e dei metodi computazionali ha reso possibile scansionare immagini istologiche ad alta risoluzione e analizzarle automaticamente utilizzando approcci basati sul machine learning. Recentemente, sono stati fatti sforzi per appaiare dati istologici e molecolari, come grandi dataset di sequenziamento dell’RNA e sequenziamento dell’intero genoma, provenienti da migliaia di campioni. Combinare queste informazioni potrebbe espandere la nostra conoscenza su come la struttura e la funzione dei tessuti variano in una popolazione e su come la variazione genetica e l’espressione genica influenzano i tessuti sani e malati.
La ricerca condotta da Francesco Cisternino, uno studente di dottorato nel laboratorio del Dr. Craig A. Glastonbury (The Glastonbury Group) presso il Centro di Genomica di Human Technopole, ha sviluppato un nuovo modello di machine learning basato sui Vision Transformers (ViT) che impara a raggruppare e segmentare automaticamente i tessuti. I ricercatori hanno combinato dati di istologia, espressione genica e variazione genetica in oltre 13.000 campioni rappresentativi di 23 tessuti umani sani provenienti da 838 donatori.
Lo studio è stato ora pubblicato su Nature Communications.
Analizzando gigapixel da Whole Slide Image , il gruppo ha trovato una significativa variabilità intra-tissutale tra i donatori e ha identificato patologie non annotate come eventi di calcificazione, incorretta classificazione e contaminazione dei tessuti. Inoltre, hanno scoperto signature di espressione genica per specifiche sottostrutture tissutali e rivelato associazioni genetiche precedentemente sconosciute.
I ricercatori hanno anche sviluppato RNAPath, un modello di machine learning che consente di prevedere e spazializzare i livelli di espressione genica solo dalle immagini istologiche H&E. RNAPath ha superato in prestazioni altri metodi concorrenti, come HE2RNA, un modello di deep learning ampiamente utilizzato per prevedere l’espressione dell’RNA-Seq dae Whole Slide Image.
In sintesi, questo studio rivela che i metodi di machine learning auto-supervisionati e gli archivi istologici possono essere utilizzati per apprendere nuove informazioni sulla patologia delle malattie e l’organizzazione dei tessuti, permettendo ai ricercatori di esplorare l’interazione tra la variabilità morfologica dei tessuti e l’espressione genica.
Craig Glastonbury, responsabile della ricerca, ha commentato: “Man mano che gli archivi istologici e i flussi di lavoro della patologia diventano digitali, crediamo che ci sia un’opportunità sostanziale per utilizzare l’apprendimento auto-supervisionato per scoprire nuove e fondamentali conoscenze sulla struttura dei tessuti, la loro funzione e la loro variabilità in una popolazione sia in soggetti sani che malati”.
Cisternino, F., Ometto, S., Chatterjee, S. et al. Self-supervised learning for characterising histomorphological diversity and spatial RNA expression prediction across 23 human tissue types. Nat Commun 15, 5906 (2024). https://doi.org/10.1038/s41467-024-50317-w
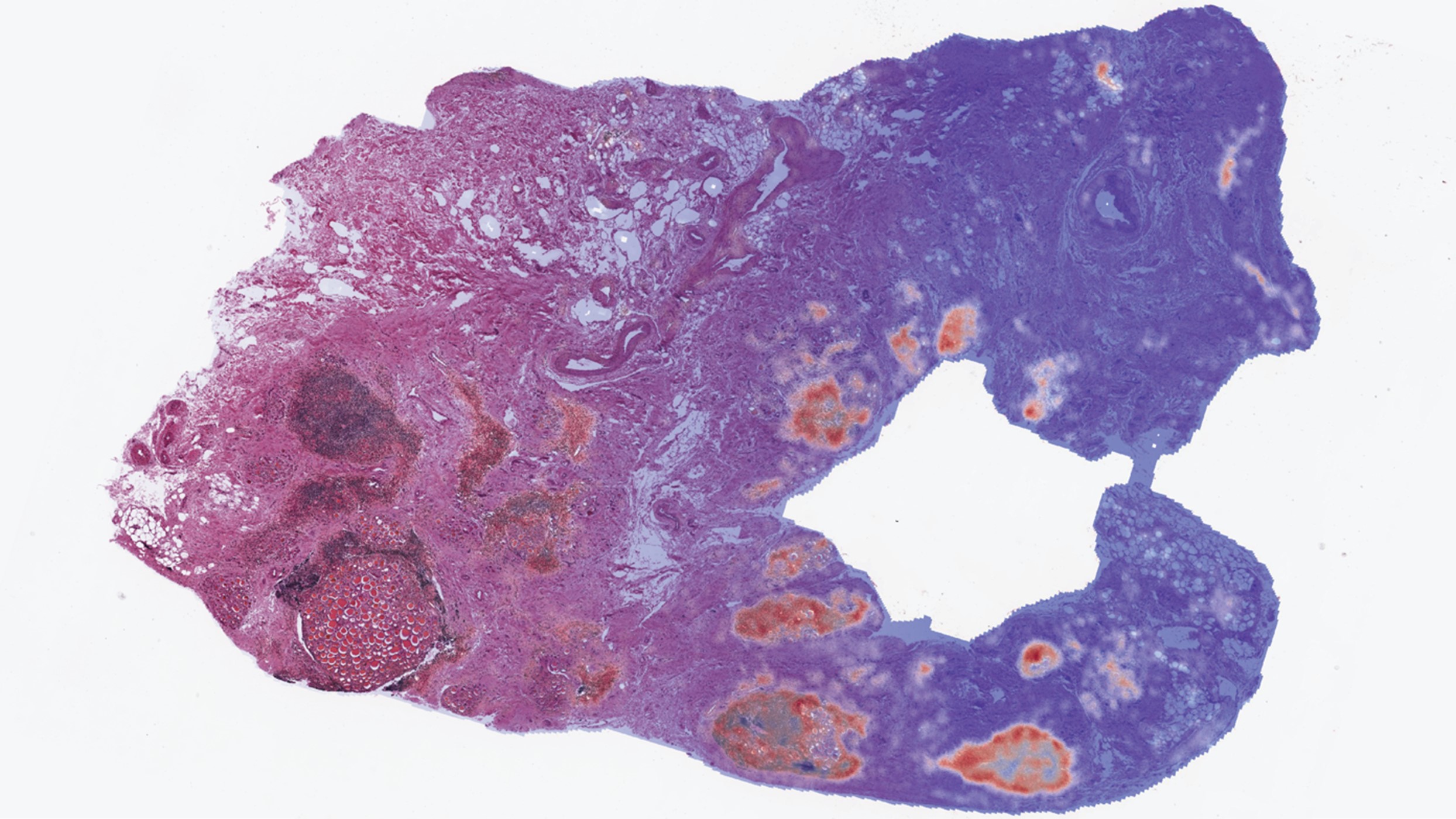
Nell’immagine: RNAPath predice la posizione spaziale dell’espressione di CD19 attraverso una sezione tessutale tiroidea H&E.