Come SNAPc recluta le RNA polimerasi umane presso i geni codificanti small nuclear RNA
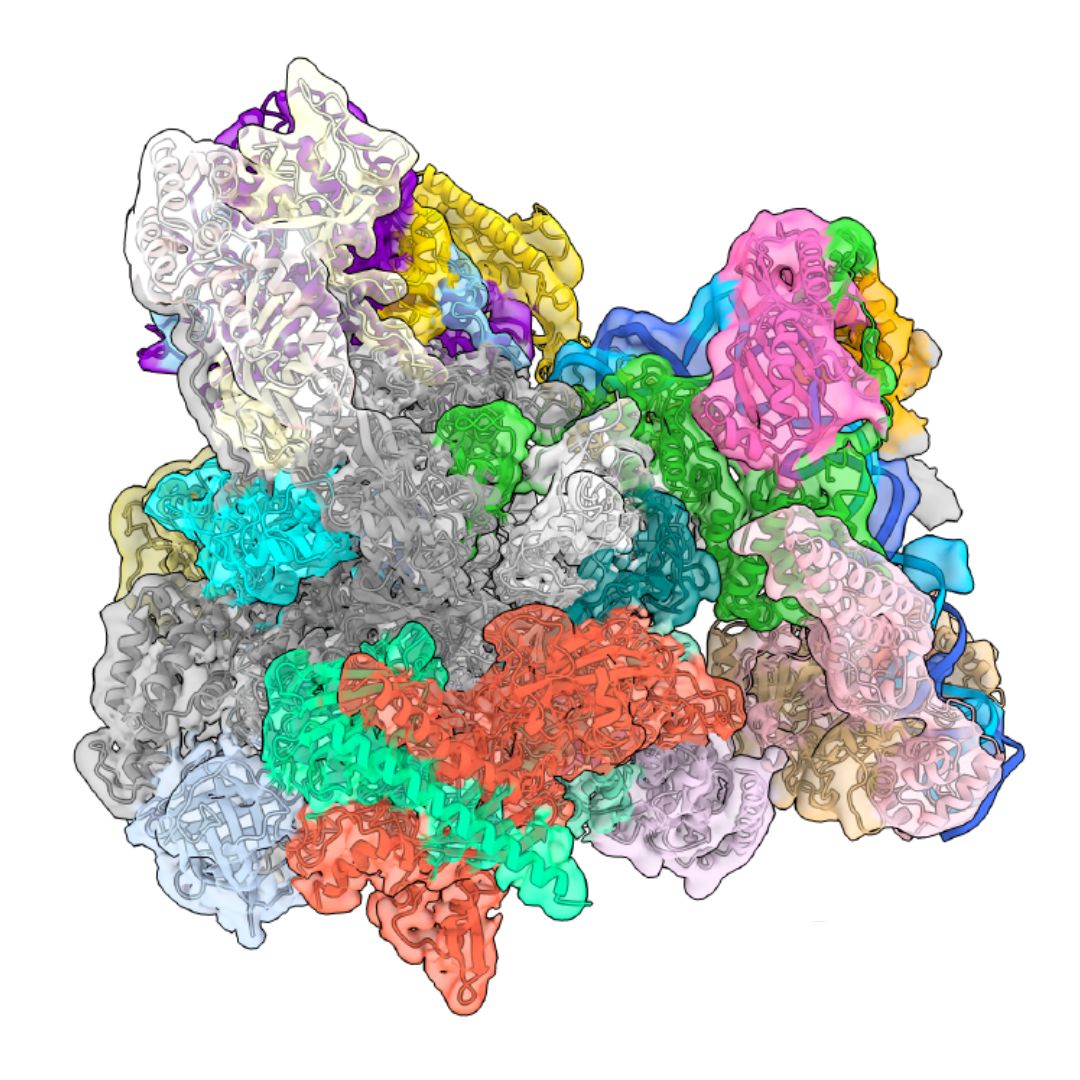
Utilizzando avanzate tecniche di microscopia crioelettronica e spettrometria di massa, i ricercatori di Human Technopole hanno svelato come SNAPc funzioni come “duplice reclutatore” per l’RNA polimerasi II e III. I risultati sono stati pubblicati su Nature Communications.
Le RNA polimerasi sono minuscole macchine molecolari che producono RNA, molecole essenziali per la vita. Tra queste, l’RNA polimerasi III (Pol III) trascrive piccoli ma fondamentali RNA come U6 small nuclear RNA (snRNA) che svolge un ruolo cruciale nello splicing dell’RNA, un processo indispensabile per la produzione di proteine funzionali. La disfunzione di Pol III è legata a diverse malattie, tra cui l’Alzheimer e il cancro.
Il reclutamento di Pol III a specifici geni U6 snRNA richiede un complesso molecolare chiamato SNAPc. Inoltre, SNAPc può reclutare anche l’RNA polimerasi II (Pol II), mediando così la sintesi di diversi tipi di snRNA. Tuttavia, il modo in cui SNAPc si lega specificamente a ciascuna polimerasi mantenendo una funzione precisa è rimasto finora poco chiaro.
Alessandro Vannini ed Ewan Ramsay – rispettivamente Head del Centro di Ricerca in Biologia Strutturale e senior staff scientist di Human Technopole – hanno sfruttato la microscopia crioelettronica (cryo-EM) per visualizzare la struttura di SNAPc in complesso con il promotore di U6 snRNA. Questo lavoro è stato condotto in collaborazione con ricercatori dell’Institute of Cancer Research di Londra (UK) e dell’Unità di Biologia Strutturale e Computazionale di EMBL (Germania).
La struttura cryo-EM di SNAPc ha rivelato che un design strutturale adattabile consente a SNAPc di reclutare diverse RNA polimerasi. SNAPc può invertire la propria orientazione a seconda del tipo di RNA polimerasi reclutata, garantendo così che la polimerasi corretta venga ingaggiata nel sito giusto. La struttura ha inoltre mostrato che la subunità SNAPC4 di SNAPc agisce come un ponte versatile, interagendo con i Pol II e Pol III in modi differenti grazie ai suoi doppi motivi di interazione . Infine, utilizzando una particolare forma di spettrometria di massa – la cross-linking mass spectrometry – in combinazione con simulazioni strutturaliottenute tramite AlphaFold, i ricercatori hanno ottenuto mappe dettagliate delle subunità SNAPC2 e SNAPC5 di SNAPc, mostrando i loro ruoli nello stabilizzare il complesso e nel regolare il legame con il DNA.
Questi risultati chiariscono i meccanismi di produzione degli snRNA a partire da specifici geni e gettano le basi per future ricerche sulla regolazione della trascrizione e sulle sue implicazioni per la salute e le malattie. Inoltre, espandono la nostra comprensione di come le cellule gestiscano con precisione compiti molecolari complessi, offrendo nuove prospettive sulla biologia fondamentale e sulle potenziali applicazioni mediche.
Shah, S.Z., Perry, T.N., Graziadei, A. et al. Structural insights into distinct mechanisms of RNA polymerase II and III recruitment to snRNA promoters. Nat Commun 16, 141 (2025). https://doi.org/10.1038/s41467-024-55553-8