La Cryo-EM fa luce sulla specificità dell’integrazione dei retrotrasposoni
I retrotrasposoni o elementi trasponibili (ET) sono parti del DNA eucariotico che possono replicarsi e incollarsi in diverse aree del genoma. Questo processo prevede la trascrizione inversa di RNA in DNA, che viene poi inserito nel genoma ospitante. Tale meccanismo di “copia e incolla” è condiviso con una classe di virus noti come retrovirus, ma non porta alla formazione di particelle infettive che possono contaminare altre cellule. Tuttavia, la replicazione e l’inserimento dei retrotrasposoni sono potenzialmente dannosi poiché possono interrompere la sequenza codificante di un gene o create siti in cui il DNA può ricombinarsi, inducendo nuove mutazioni nel genoma.
I retrotrasposoni che presentano alle estremità delle lunghe sequenze ripetute terminali (LTR) rappresentano la principale classe di ET nel genoma eucariotico. Ty3/Gypsy è un LTR ben caratterizzato nel lievito Saccharomyces cerevisiae, ed è noto per inserirsi a monte dei geni dell’RNA transfer (tRNA) essenziali per la sintesi delle proteine. I fattori di trascrizione generali TFIIB e TFIIC mediano l’inserimento di Ty3 nei geni tRNA, conferendo rispettivamente specificità di targeting e direzionalità dell’integrazione. Tuttavia, sappiamo poco di come si ottenga tale specificità.
Alessandro Vannini, Head del Centro di Biologia Strutturale di Human Technopole, ed il tuo team hanno utilizzato la microscopia crio-elettronica (Cryo-EM), una tecnica di imaging particolarmente avanzata, per caratterizzare l’interazione di Ty3, TFIIB e TFIIC con i geni bersaglio di tRNA e rivelare i meccanismi molecolari alla base della specificità dell’integrazione di Ty3. I risultati della loro ricerca sono stati pubblicati su Nature Communications.
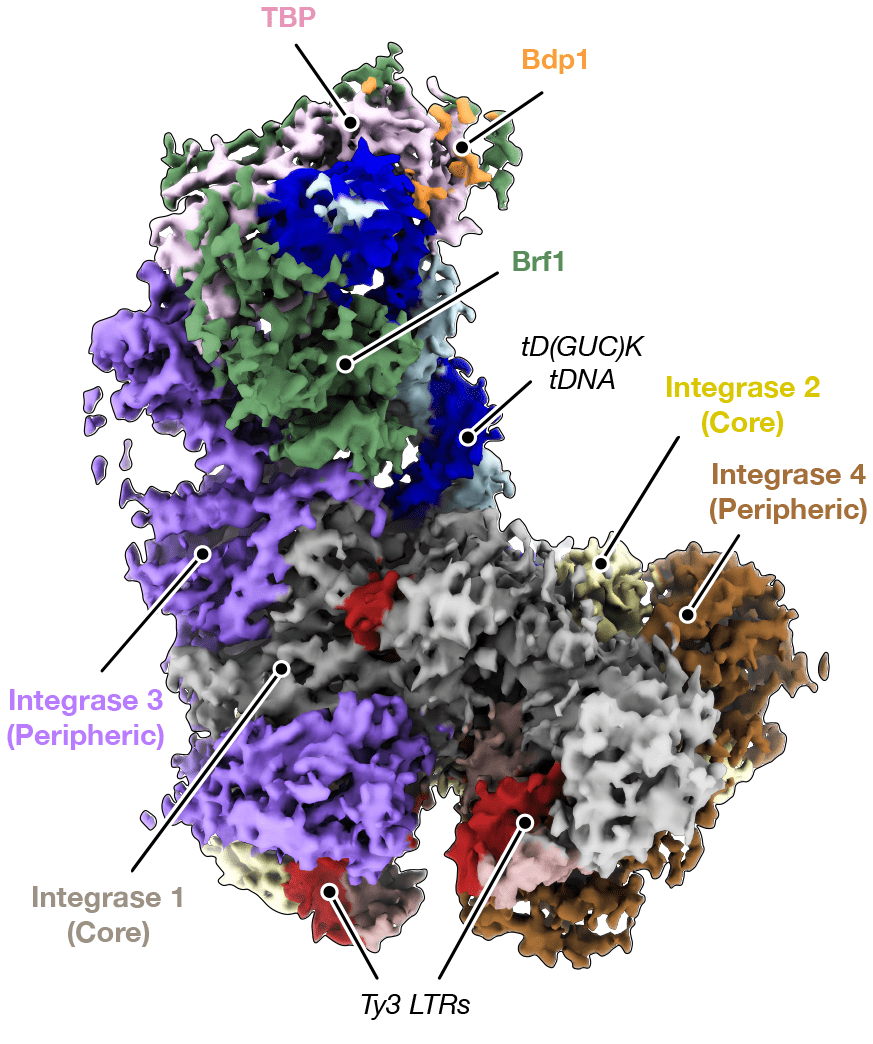
I ricercatori hanno purificato il macchinario molecolare del retrotrasposone Ty3 – composto da una trascrittasi inversa ed un’integrasi, di seguito denominata intasoma – e hanno ricostituito l’intasoma Ty3 legato a TFIIB o in complesso con TFIIB e DNA bersaglio, utilizzando la Cryo-EM.
Hanno così scoperto che il dominio C-terminale (CTD) e un cromodominio (CHD) precedentemente non caratterizzato dell’integrasi Ty3, specificavano la posizione dell’inserimento del gene Ty3 a monte del promotore del gene del tRNA bersaglio. Inoltre, per il reclutamento dell’intasoma Ty3 da parte di TFIIB è risultata necessaria una lunga regione linker tra il CTD e il dominio catalitico (CCD) dell’integrasi Ty3. In particolare, la modalità di legame dell’intasoma Ty3 a TFIIIB assomigliava a quella dell’intasoma del virus dell’immunodeficienza umana (HIV) al gene del fattore di crescita derivato dall’epitelio del cristallino (LEDGF/p75). Questo parallelo suggerisce che, nonostante siano evolutivamente distanti, gli intasomi di HIV e Ty3 utilizzano regioni conservate alla loro periferia per localizzarsi nel DNA dell’ospite.
Per studiare come avviene l’integrazione del retrotrasposone Ty3, i ricercatori hanno confrontato le strutture Cryo-EM della RNA Polimerasi III (Pol III) – responsabile della trascrizione del gene tRNA – e dell’intasoma Ty3 reclutato da TFIIIB con un gene trascritto da Pol III e hanno dimostrato che il loro legame al DNA bersaglio richiede regioni equivalenti, che prevengono la co-occorrenza della trascrizione del tRNA e l’integrazione di Ty3.
In sintesi, Vannini e il suo gruppo hanno rivelato le basi strutturali alla base della specificità dell’integrazione di Ty3 nei geni trascritti da Pol III e hanno mostrato come queste caratteristiche siano conservate in ET evolutivamente distanti.