Vannini Group
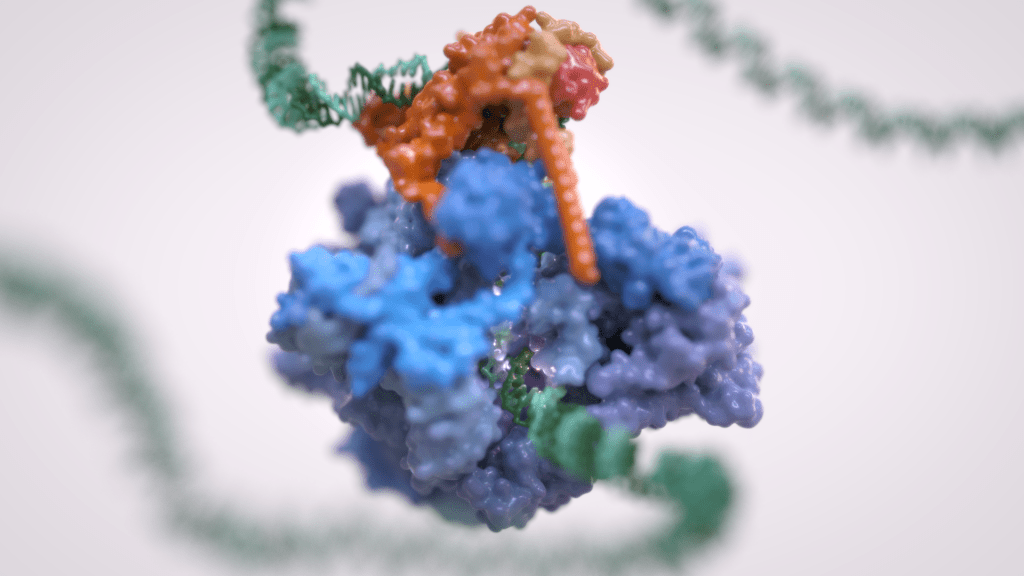
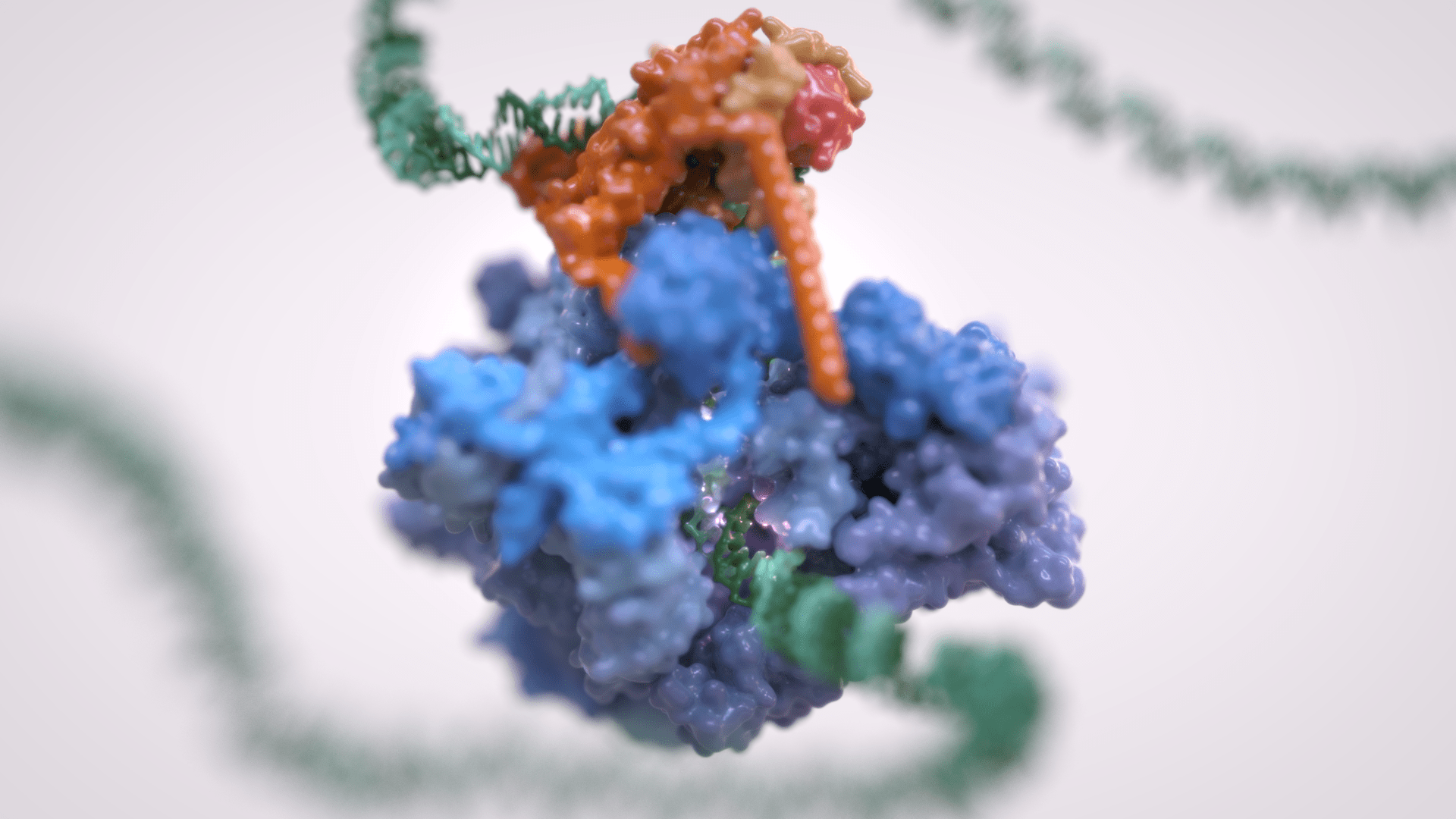
La trascrizione genica è il primo passaggio che regola l’espressione dell’informazione genetica codificata in un genoma, ed è alla base della differenziazione cellulare e dello sviluppo dell’organismo. La trascrizione genica eucariotica avviene nel contesto di genomi altamente strutturati e organizzati e coordina numerosi eventi che si verificano nel nucleo. La trascrizione eucariotica si basa su tre diverse RNA polimerasi: L’RNA polimerasi I (Pol I) trascrive l’RNA ribosomiale, l’RNA polimerasi II (Pol II) sintetizza gli RNA messaggeri e l’RNA polimerasi III (Pol III) produce RNA brevi e non tradotti, compreso l’intero pool di tRNA, che sono essenziali per la crescita cellulare.
Per molto tempo si è ritenuto che solo Pol II fosse regolata e che Pol I e Pol III non richiedessero tale controllo, essendo dedicati a geni housekeeping. Tuttavia, è ora chiaro come la trascrizione dell’RNA polimerasi III sia strettamente regolata e sia un fattore determinante per la crescita di un organismo. La deregolazione di Pol III è stata osservata in varie forme di cancro e mutazioni genetiche a carico di Pol III causano gravi malattie neurodegenerative.
Inoltre, Pol III e i suoi fattori associati svolgono un ruolo fondamentale nella struttura e nell’organizzazione del genoma. Questi “ruoli extra-trascrizionali” sono svolti attraverso interazioni con altri componenti cellulari quali i transposoni, i complessi SMC (dall’inglese Structural Maintenance of Chromosomes) e i rimodellatori specifici della cromatina.
Il Vannini Group utilizza un approccio di Biologia Strutturale Integrativa, che combina analisi di crio-microscopia elettronica all’avanguardia, dati di diffrazione dei raggi X, cross-linking e spettrometria di massa nativa. Integriamo i dati strutturali con le tecniche di biologia molecolare e cellulare per ottenere una visione globale di questi processi fondamentali e di come la loro errata regolazione possa condurre a malattie oncologiche e neurodegenerative.
Membri del gruppo
-
 Alessandro Vannini
Alessandro Vannini
Head of Structural Biology Research Centre -
 Alessandro Borsellini
Alessandro Borsellini
Postdoc -
 Giacomo Ettore Casale
Giacomo Ettore Casale
Postdoc -
 Valentina Cecatiello
Valentina Cecatiello
Senior Technician -
 Sebastian Chamera
Sebastian Chamera
Postdoc -
 Fabiola Iommazzo
Fabiola Iommazzo
PhD Student -
 Thomas Noé Perry
Thomas Noé Perry
Postdoc -
 Fabio Pessina
Fabio Pessina
Senior Technician -
 Mariavittoria Pizzinga
Mariavittoria Pizzinga
Postdoc -
 Ewan Ramsay
Ewan Ramsay
Senior Staff Scientist -
 Ankit Roy
Ankit Roy
PhD Student -
 Syed Zawar Shah
Syed Zawar Shah
PhD Student
Pubblicazioni
-
07/2020 - Nature Metabolism
A micronutrient with major effects on cancer cell viability
Selenium is a micronutrient essential for the generation of selenoproteins, which function predominantly by detoxifying cellular reactive oxygen species. In this issue, Carlisle et al. describe a novel mechanism whereby perturbing selenium utilization via inhibition of SEPHS2, a component of the selenocysteine-biosynthesis pathway, results in selenide poisoning and cancer cell death.
-
07/2020 - Molecular Cell
Human Condensin I and II Drive Extensive ATP-Dependent Compaction of Nucleosome-Bound DNA
Structural maintenance of chromosomes (SMC) complexes are essential for genome organization from bacteria to humans, but their mechanisms of action remain poorly understood. Here, we characterize human SMC complexes condensin I and II and unveil the architecture of the human condensin II complex, revealing two putative DNA-entrapment sites. Using single-molecule imaging, we demonstrate that both […]
-
06/2020 - Cell
Hybrid Gene Origination Creates Human-Virus Chimeric Proteins during Infection
RNA viruses are a major human health threat. The life cycles of many highly pathogenic RNA viruses like influenza A virus (IAV) and Lassa virus depends on host mRNA, because viral polymerases cleave 5′-m7G-capped host transcripts to prime viral mRNA synthesis (“cap-snatching”). We hypothesized that start codons within cap-snatched host transcripts could generate chimeric human-viral […]
-
06/2020 - Nature Communications
DNA origami-based single-molecule force spectroscopy elucidates RNA Polymerase III pre-initiation complex stability
The TATA-binding protein (TBP) and a transcription factor (TF) IIB-like factor are important constituents of all eukaryotic initiation complexes. The reason for the emergence and strict requirement of the additional initiation factor Bdp1 in the RNA polymerase (RNAP) III system, however, remained elusive. A poorly studied aspect in this context is the effect of DNA […]
-
11/2019 - Molecular Cell
TFIIIC Binding to Alu Elements Controls Gene Expression via Chromatin Looping and Histone Acetylation
How repetitive elements, epigenetic modifications, and architectural proteins interact ensuring proper genome expression remains poorly understood. Here, we report regulatory mechanisms unveiling a central role of Alu elements (AEs) and RNA polymerase III transcription factor C (TFIIIC) in structurally and functionally modulating the genome via chromatin looping and histone acetylation. Upon serum deprivation, a subset […]