Vannini Group
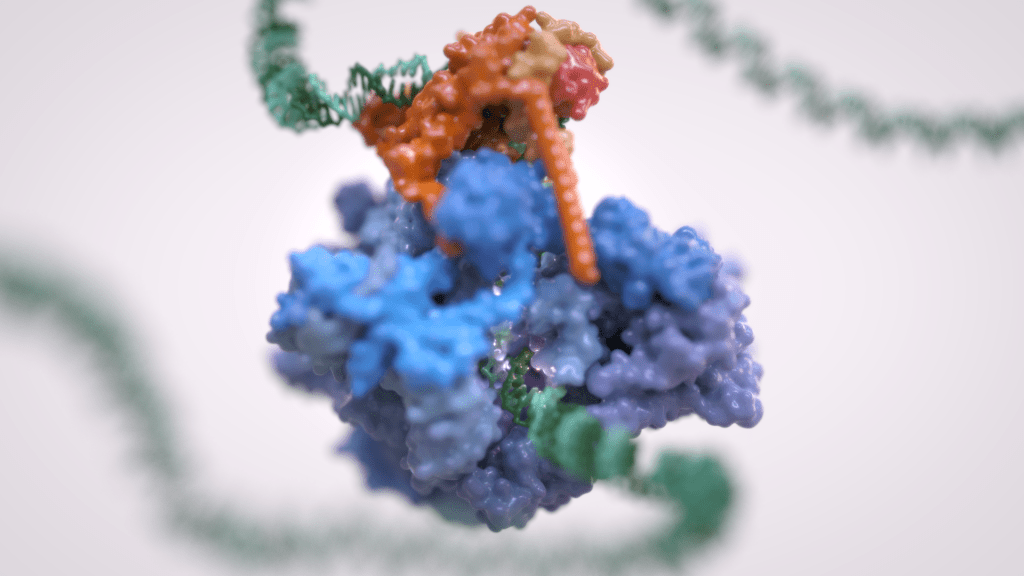
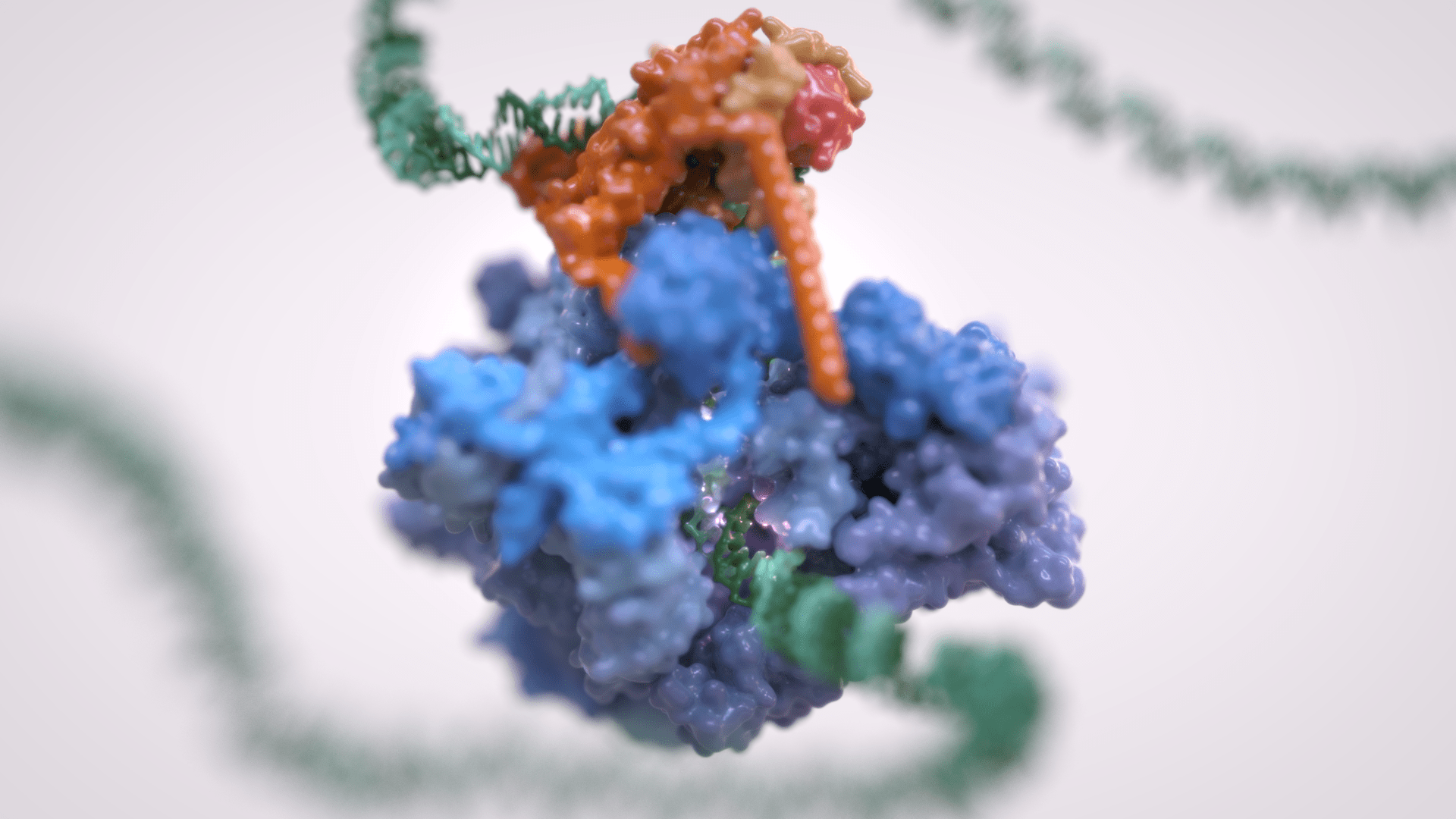
La trascrizione genica è il primo passaggio che regola l’espressione dell’informazione genetica codificata in un genoma, ed è alla base della differenziazione cellulare e dello sviluppo dell’organismo. La trascrizione genica eucariotica avviene nel contesto di genomi altamente strutturati e organizzati e coordina numerosi eventi che si verificano nel nucleo. La trascrizione eucariotica si basa su tre diverse RNA polimerasi: L’RNA polimerasi I (Pol I) trascrive l’RNA ribosomiale, l’RNA polimerasi II (Pol II) sintetizza gli RNA messaggeri e l’RNA polimerasi III (Pol III) produce RNA brevi e non tradotti, compreso l’intero pool di tRNA, che sono essenziali per la crescita cellulare.
Per molto tempo si è ritenuto che solo Pol II fosse regolata e che Pol I e Pol III non richiedessero tale controllo, essendo dedicati a geni housekeeping. Tuttavia, è ora chiaro come la trascrizione dell’RNA polimerasi III sia strettamente regolata e sia un fattore determinante per la crescita di un organismo. La deregolazione di Pol III è stata osservata in varie forme di cancro e mutazioni genetiche a carico di Pol III causano gravi malattie neurodegenerative.
Inoltre, Pol III e i suoi fattori associati svolgono un ruolo fondamentale nella struttura e nell’organizzazione del genoma. Questi “ruoli extra-trascrizionali” sono svolti attraverso interazioni con altri componenti cellulari quali i transposoni, i complessi SMC (dall’inglese Structural Maintenance of Chromosomes) e i rimodellatori specifici della cromatina.
Il Vannini Group utilizza un approccio di Biologia Strutturale Integrativa, che combina analisi di crio-microscopia elettronica all’avanguardia, dati di diffrazione dei raggi X, cross-linking e spettrometria di massa nativa. Integriamo i dati strutturali con le tecniche di biologia molecolare e cellulare per ottenere una visione globale di questi processi fondamentali e di come la loro errata regolazione possa condurre a malattie oncologiche e neurodegenerative.
Membri del gruppo
-
 Alessandro Vannini
Alessandro Vannini
Head of Structural Biology Research Centre -
 Alessandro Borsellini
Alessandro Borsellini
Postdoc -
 Giacomo Ettore Casale
Giacomo Ettore Casale
Postdoc -
 Valentina Cecatiello
Valentina Cecatiello
Senior Technician -
 Sebastian Chamera
Sebastian Chamera
Postdoc -
 Fabiola Iommazzo
Fabiola Iommazzo
PhD Student -
 Thomas Noé Perry
Thomas Noé Perry
Postdoc -
 Fabio Pessina
Fabio Pessina
Senior Technician -
 Mariavittoria Pizzinga
Mariavittoria Pizzinga
Postdoc -
 Ewan Ramsay
Ewan Ramsay
Senior Staff Scientist -
 Ankit Roy
Ankit Roy
PhD Student -
 Syed Zawar Shah
Syed Zawar Shah
PhD Student
Pubblicazioni
-
12/2015 - Cell
Redox Signaling by the RNA Polymerase III TFIIB-Related Factor Brf2
TFIIB-related factor 2 (Brf2) is a member of the family of TFIIB-like core transcription factors. Brf2 recruits RNA polymerase (Pol) III to type III gene-external promoters, including the U6 spliceosomal RNA and selenocysteine tRNA genes. Found only in vertebrates, Brf2 has been linked to tumorigenesis but the underlying mechanisms remain elusive. We have solved crystal […]
-
09/2012 - Biochim Biophys Acta
A structural perspective on RNA polymerase I and RNA polymerase III transcription machineries
RNA polymerase I and III are responsible for the bulk of nuclear transcription in actively growing cells and their activity impacts the cellular biosynthetic capacity. As a consequence, RNA polymerase I and III deregulation has been directly linked to cancer development. The complexity of RNA polymerase I and III transcription apparatuses has hampered their structural […]
-
02/2012 - Molecular Cell
Conservation between the RNA polymerase I, II, and III transcription initiation machineries
Recent studies of the three eukaryotic transcription machineries revealed that all initiation complexes share a conserved core. This core consists of the RNA polymerase (I, II, or III), the TATA box-binding protein (TBP), and transcription factors TFIIB, TFIIE, and TFIIF (for Pol II) or proteins structurally and functionally related to parts of these factors (for […]
-
10/2010 - Cell
Molecular basis of RNA polymerase III transcription repression by Maf1
RNA polymerase III (Pol III) transcribes short RNAs required for cell growth. Under stress conditions, the conserved protein Maf1 rapidly represses Pol III transcription. We report the crystal structure of Maf1 and cryo-electron microscopic structures of Pol III, an active Pol III-DNA-RNA complex, and a repressive Pol III-Maf1 complex. Binding of DNA and RNA causes […]
-
10/2010 - Nucleic Acids Res
The archaeo-eukaryotic primase of plasmid pRN1 requires a helix bundle domain for faithful primer synthesis
The plasmid pRN1 encodes for a multifunctional replication protein with primase, DNA polymerase and helicase activity. The minimal region required for primase activity encompasses amino-acid residues 40-370. While the N-terminal part of that minimal region (residues 47-247) folds into the prim/pol domain and bears the active site, the structure and function of the C-terminal part […]