Vannini Group
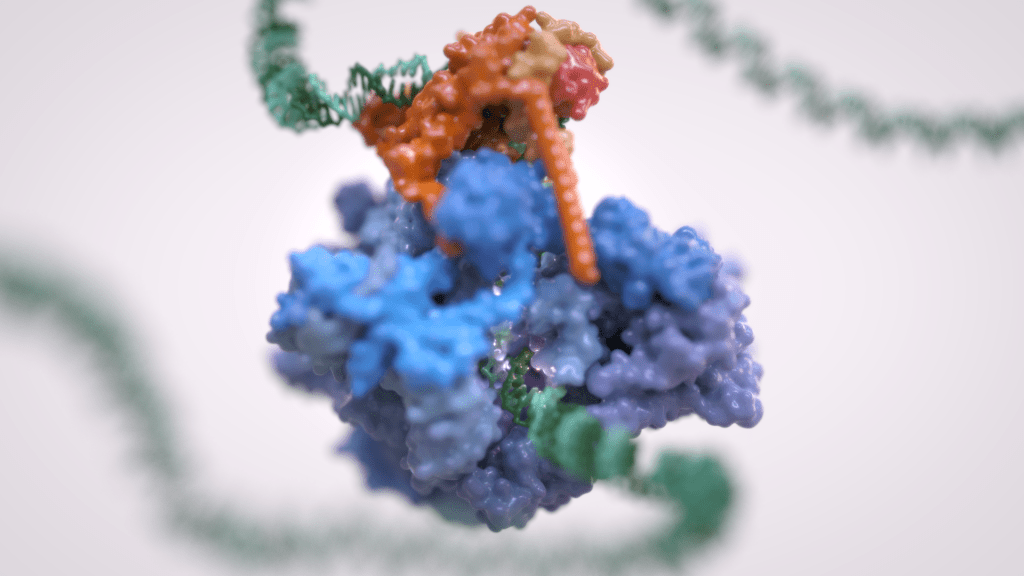
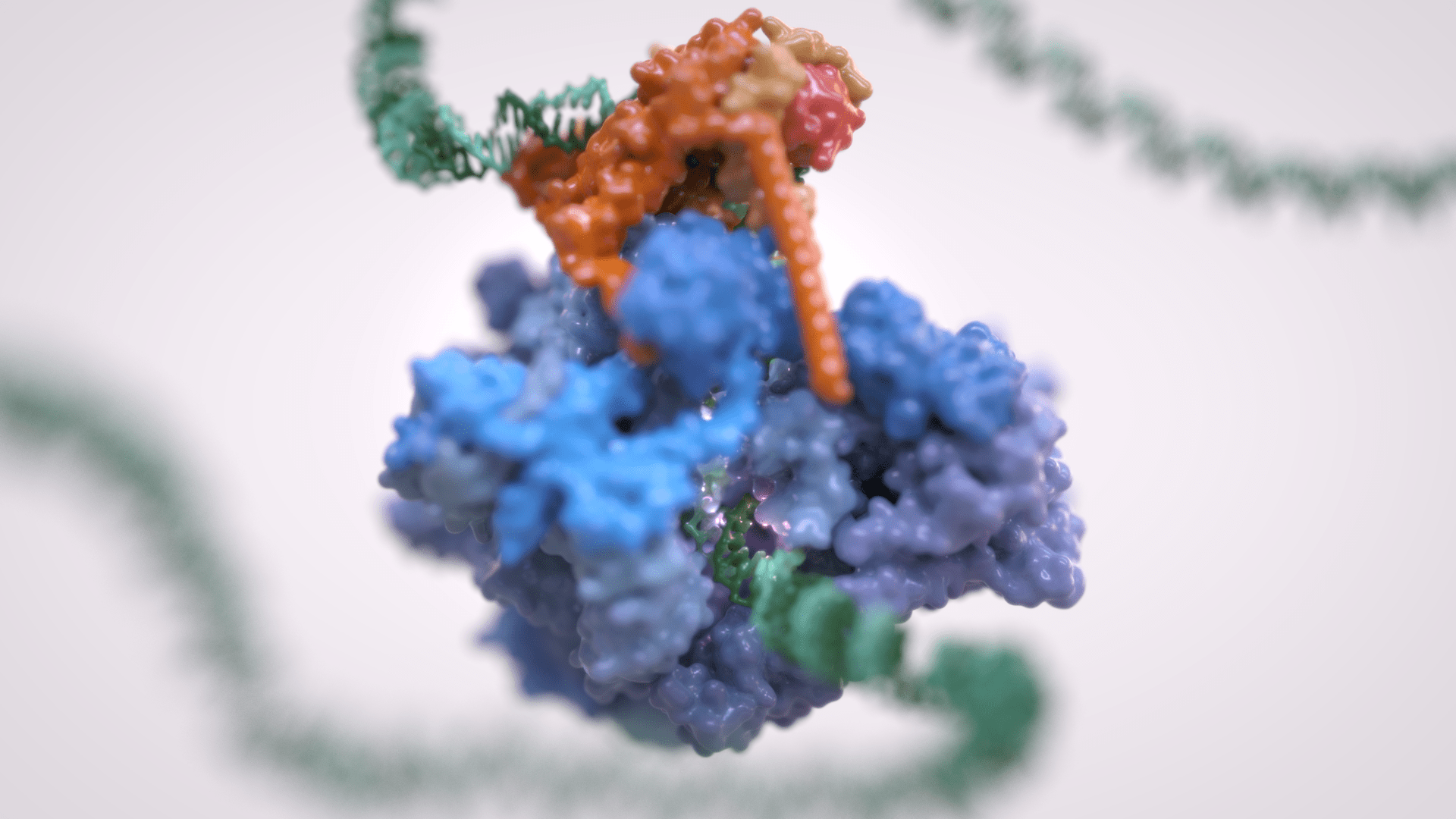
La trascrizione genica è il primo passaggio che regola l’espressione dell’informazione genetica codificata in un genoma, ed è alla base della differenziazione cellulare e dello sviluppo dell’organismo. La trascrizione genica eucariotica avviene nel contesto di genomi altamente strutturati e organizzati e coordina numerosi eventi che si verificano nel nucleo. La trascrizione eucariotica si basa su tre diverse RNA polimerasi: L’RNA polimerasi I (Pol I) trascrive l’RNA ribosomiale, l’RNA polimerasi II (Pol II) sintetizza gli RNA messaggeri e l’RNA polimerasi III (Pol III) produce RNA brevi e non tradotti, compreso l’intero pool di tRNA, che sono essenziali per la crescita cellulare.
Per molto tempo si è ritenuto che solo Pol II fosse regolata e che Pol I e Pol III non richiedessero tale controllo, essendo dedicati a geni housekeeping. Tuttavia, è ora chiaro come la trascrizione dell’RNA polimerasi III sia strettamente regolata e sia un fattore determinante per la crescita di un organismo. La deregolazione di Pol III è stata osservata in varie forme di cancro e mutazioni genetiche a carico di Pol III causano gravi malattie neurodegenerative.
Inoltre, Pol III e i suoi fattori associati svolgono un ruolo fondamentale nella struttura e nell’organizzazione del genoma. Questi “ruoli extra-trascrizionali” sono svolti attraverso interazioni con altri componenti cellulari quali i transposoni, i complessi SMC (dall’inglese Structural Maintenance of Chromosomes) e i rimodellatori specifici della cromatina.
Il Vannini Group utilizza un approccio di Biologia Strutturale Integrativa, che combina analisi di crio-microscopia elettronica all’avanguardia, dati di diffrazione dei raggi X, cross-linking e spettrometria di massa nativa. Integriamo i dati strutturali con le tecniche di biologia molecolare e cellulare per ottenere una visione globale di questi processi fondamentali e di come la loro errata regolazione possa condurre a malattie oncologiche e neurodegenerative.
Membri del gruppo
-
 Alessandro Vannini
Alessandro Vannini
Head of Structural Biology Research Centre -
 Alessandro Borsellini
Alessandro Borsellini
Postdoc -
 Giacomo Ettore Casale
Giacomo Ettore Casale
Postdoc -
 Valentina Cecatiello
Valentina Cecatiello
Senior Technician -
 Sebastian Chamera
Sebastian Chamera
Postdoc -
 Fabiola Iommazzo
Fabiola Iommazzo
PhD Student -
 Thomas Noé Perry
Thomas Noé Perry
Postdoc -
 Fabio Pessina
Fabio Pessina
Senior Technician -
 Mariavittoria Pizzinga
Mariavittoria Pizzinga
Postdoc -
 Ewan Ramsay
Ewan Ramsay
Senior Staff Scientist -
 Ankit Roy
Ankit Roy
PhD Student -
 Syed Zawar Shah
Syed Zawar Shah
PhD Student
Pubblicazioni
-
05/2008 - Analytical Chemistry
Multiplexed proteomics mapping of yeast RNA polymerase II and III allows near-complete sequence coverage and reveals several novel phosphorylation sites
The multisubunit RNA polymerases (Pols) II and III synthesize mainly eukaryotic mRNAs and tRNAs, respectively. Pol II and Pol III are protein complexes consisting of 12 and 17 subunits. Here we analyzed both yeast Pol II and Pol III by multiplexed mass spectrometric analysis using various proteases and both collision induced and electron transfer dissociation. […]
-
01/2008 - Annu Rev Biophys
Structure of eukaryotic RNA polymerases
The eukaryotic RNA polymerases Pol I, Pol II, and Pol III are the central multiprotein machines that synthesize ribosomal, messenger, and transfer RNA, respectively. Here we provide a catalog of available structural information for these three enzymes. Most structural data have been accumulated for Pol II and its functional complexes. These studies have provided insights […]
-
10/2007 - Structure
Structural biology of RNA polymerase III: mass spectrometry elucidates subcomplex architecture
RNA polymerases (Pol) II and III synthesize eukaryotic mRNAs and tRNAs, respectively. The crystal structure of the 12 subunit Pol II is known, but only limited structural information is available for the 17 subunit Pol III. Using mass spectrometry (MS), we correlated masses of Pol II complexes with the Pol II structure. Analysis of Pol […]
-
09/2007 - EMBO Rep
Substrate binding to histone deacetylases as shown by the crystal structure of the HDAC8-substrate complex
Histone deacetylases (HDACs)-an enzyme family that deacetylates histones and non-histone proteins-are implicated in human diseases such as cancer, and the first-generation of HDAC inhibitors are now in clinical trials. Here, we report the 2.0 A resolution crystal structure of a catalytically inactive HDAC8 active-site mutant, Tyr306Phe, bound to an acetylated peptidic substrate. The structure clarifies […]
-
10/2004 - Proc Natl Acad Sci U S A
Crystal structure of a eukaryotic zinc-dependent histone deacetylase, human HDAC8, complexed with a hydroxamic acid inhibitor
Histone deacetylases (HDACs) are a family of enzymes involved in the regulation of gene expression, DNA repair, and stress response. These processes often are altered in tumors, and HDAC inhibitors have had pronounced antitumor activity with promising results in clinical trials. Here, we report the crystal structure of human HDAC8 in complex with a hydroxamic […]