Immagini di microscopia più chiare nella biologia spaziale
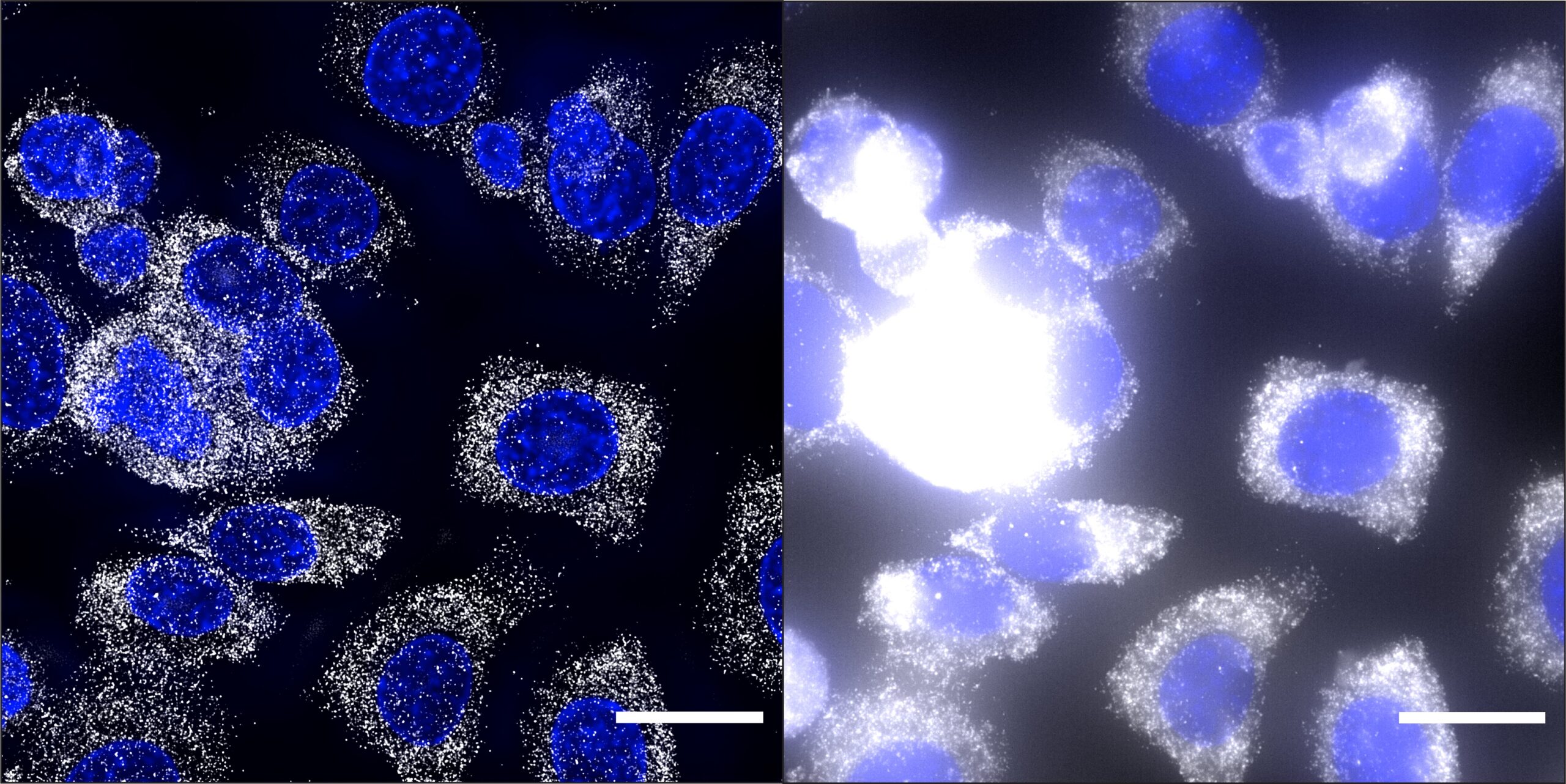
Un team internazionale di scienziati, guidato dai ricercatori di HT Magda Bienko e Nicola Crosetto, ha sviluppato un software open-source per la deconvoluzione di immagini di microscopia a fluorescenza a campo largo e scansioni di tessuti di grandi dimensioni. Questo nuovo strumento aumenta le informazioni ottenute con i metodi di omica spaziale basati sulla microscopia a fluorescenza.
Risoluzione è un termine che i microscopisti usano per indicare la capacità di distinguere i dettagli in un campione sottoposto a imaging. Più alta è la risoluzione di una micrografia, più chiari e nitidi sono i suoi elementi fini. La risoluzione dell’immagine dipende strettamente dalle caratteristiche del microscopio, come il tipo di lente, la lunghezza d’onda della luce utilizzata per l’imaging e la rimozione del segnale fuori fuoco. I microscopi confocali e a super-risoluzione garantiscono un’alta risoluzione, ma sono costosi e richiedono operatori altamente qualificati.
La deconvoluzione è una tecnica di elaborazione delle immagini che aumenta il contrasto e la risoluzione di immagini, consentendo di distinguere singoli loci del DNA o singole molecole di RNA o proteine in singole cellule all’interno di tessuti e organi, utilizzando microscopi ottici convenzionali e metodi omici spaziali basati sulla microscopia a fluorescenza. Tuttavia, i software di deconvoluzione disponibili in commercio presentano diversi limiti, che comprendono costi elevati, incapacità di elaborare immagini contenenti un’alta densità di segnali, e nonché la necessità di una formazione pratica approfondita e di computer potenti.
Per superare questi inconvenienti, Magda Bienko e Nicola Crosetto, ricercatori di Genomica dello Human Technopole, insieme a un team di ricercatori svedesi, spagnoli e statunitensi, hanno sviluppato un nuovo software di deconvoluzione chiamato Deconwolf, disponibile gratuitamente e facile da usare.
The results of the research are published in Nature Methods.
Il team ha testato Deconwolf su immagini di microscopia a fluorescenza che mostrano punti di fluorescenza al limite (o quasi) della diffrazione (immagini di DNA e RNA FISH) o immagini derivanti da omica spaziale contenenti un’alta densità di segnali, come quele prodotte dalla trascrittomica spaziale in situ. Hanno dimostrato che Deconwolf ha migliorato la risoluzione e la quantità di informazioni nelle immagini ottenute con questi metodi ad alto rendimento. Inoltre, ha superato le prestazioni di due tra i software di deconvoluzione commerciali più utilizzati.
Deconwolf può essere scaricato gratuitamente dal sito https://deconwolf.fht.org/ (sviluppo web di Riccardo De Lucia, Biologia Computazionale di Human Technopole) e funziona su computer portatili con configurazioni standard.
“Deconwolf rappresenta uno strumento potenzialmente trasformativo che può essere adottato in molti campi delle scienze della vita e della biomedicina per aumentare la quantità di informazioni biologiche recuperate dai dati di imaging generati da tecniche che si basano sulla rilevazione della fluorescenza utilizzando la microscopia a largo campo e metodi omici a risoluzione spaziale basati sull’imaging che generano segnali limitati dalla (quasi) diffrazione. Prevediamo quindi che Deconwolf democratizzerà l’uso della deconvoluzione in microscopia e troverà numerose applicazioni sia nella ricerca di base sia in diagnostica“, affermano gli autori.
Wernersson, E., Gelali, E., Girelli, G. et al. Deconwolf enables high-performance deconvolution of widefield fluorescence microscopy images. Nat Methods (2024). https://doi.org/10.1038/s41592-024-02294-7