Un nuovo approccio per studiare l’impatto della ricombinazione dei retrotrasposoni sul genoma umano
La ricombinazione dei retrotrasposoni ha effetti sulla stabilità e sull’evoluzione del genoma. Tuttavia, l’importanza di questo processo e il suo impatto sul genoma delle cellule somatiche umane sono poco conosciuti. Giovanni Pascarella e Piero Carninci presentano un nuovo metodo che consente di studiare, nella sua globalità, la diversità genomica indotta dalla ricombinazione dei retrotrasposoni nei tessuti sani e malati.
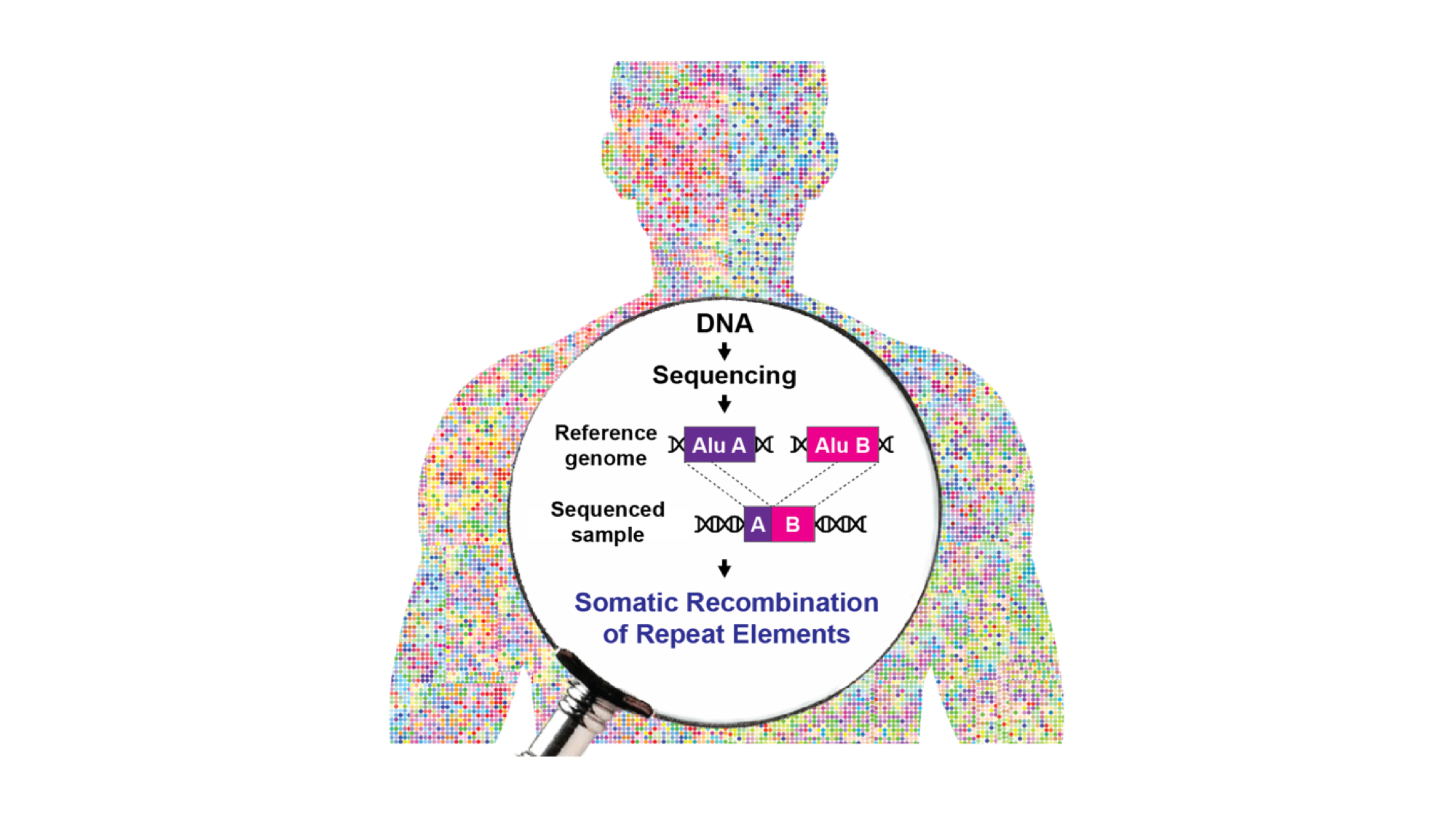
Il DNA contenuto nel nucleo di ogni cellula del nostro corpo è costantemente sottoposto a stimoli endogeni ed esogeni che possono provocare la rottura dei due filamenti della doppia elica (in inglese, double strand breaks o DSB). Queste lesioni del DNA mettono a rischio l’integrità del genoma e devono quindi essere riparate tempestivamente per evitare l’innesco di vie apoptotiche. Una delle principali vie di riparazione dei DSB del DNA è la ricombinazione omologa, che può avvenire i) tra due loci identici su cromatidi gemelli o cromosomi omologhi, portando alla risoluzione neutrale della rottura, oppure ii) tra loci non allelici (ricombinazione omologa non allelica, NAHR). Quest’ultimo processo può generare diversità genetica e contribuire all’evoluzione del genoma, ma può anche essere dannoso per l’organismo. Il genoma umano ospita milioni di copie di retrotrasposoni o elementi trasponibili; LINE-1 (L1) e Alu sono i più numerosi e rappresentano circa un terzo dell’intero genoma. A causa dell’elevato numero di copie e identità, questi elementi trasponibili sono stati spesso coinvolti in eventi NAHR che portano a diversi tipi di riarrangiamenti del DNA nel cancro e nelle malattie genetiche. Tuttavia, l’impatto della NAHR sulla variazione del genoma delle cellule somatiche ed il contributo dei diversi retrotrasposoni al riarrangiamento genomico NAHR-dipendente rimangono poco conosciuti.
Per studiare questi problemi, Piero Carninci – Responsabile del Centro di Genomica di Human Technopole, Team Leader del Laboratory for Transcriptome Technology, e vicedirettore del Centro RIKEN per le Scienze Mediche Integrative (IMS) di Yokohama (Giappone) – e Giovanni Pascarella – ricercatore dell’IMS – hanno creato un protocollo che prevede l’arricchimento delle sequenze di retrotrasposoni (“capture-seq”) e la loro successiva analisi mediante un avanzato approccio bioinformatico (TE-reX) per studiare gli eventi NAHR somatici negli elementi Alu e L1 in tessuti post-mortem di diversa derivazione embrionale, compresi il fegato, i reni e tre regioni cerebrali corticali di dieci donatori sani. I risultati dello studio sono ora pubblicati su Cell.
I ricercatori hanno scoperto che il numero di eventi NAHR associati alla ricombinazione di Alu e L1 é maggiore nel fegato e nel rene rispetto al tessuto cerebrale. Nel cervello hanno invece trovato un alto numero di eventi NAHR intracromosomici e di ricombinazione tra retrotrasposoni vicini tra loro, che si verificano preferibilmente nei cromosomi più corti. Inoltre, gli scienziati hanno visto che la ricombinazione intracromosomica di ripetizioni invertite vicine rappresenta una fonte di instabilità genomica a seconda del tessuto analizzato. Infine, la ricombinazione di L1 e Alu é arricchita nei centrosomi umani, e copie di Alu particolarmente attive per NAHR costituiscono hotspots di ricombinazione in oncogeni e geni soppressori di tumori elencati nel database COSMIC. Ma quali sono l’origine e il ruolo fisiologico della ricombinazione tessuto-specifica di Alu e L1? Pascarella e colleghi hanno risposto a questa domanda analizzando gli eventi ricombinazione somatica in vitro, durante il differenziamento di cellule staminali pluripotenti (iPSC) in neuroni (iNEU). In accordo con i risultati ottenuti nel tessuto cerebrale, le iNEU hanno mostrato tassi di ricombinazione di coppie Alu e L1 prossimali più elevati e una marcata correlazione inversa tra la lunghezza del cromosoma e i tassi di ricombinazione intracromosomica rispetto alle iPSC, suggerendo così che la ricombinazione delle sequenze Alu e L1 può essere legata a riarrangiamenti del DNA durante la differenziazione neuronale precoce e lo sviluppo cerebrale.
“Le nostre analisi della ricombinazione nelle iPSC e nei neuroni differenziati suggeriscono che gli specifici profili di ricombinazione osservati nei campioni post-mortem possono essere generati nelle cellule progenitrici durante le prime fasi dello sviluppo. È comunque una questione aperta se i profili di ricombinazione specifici del tessuto siano una causa o una conseguenza della determinazione del destino cellulare”, afferma Giovanni Pascarella, primo autore e coordinatore dello studio.
Successivamente, il team ha analizzato la NAHR somatica tessuto-specifica di Alu e L1 in due condizioni patologiche, il Parkinson (PD) sporadico e la malattia di Alzheimer (AD), tracciando il profilo della NAHR in campioni post-mortem da pazienti con PD e AD. Queste analisi hanno rivelato che gli eventi NAHR sono arricchiti nei geni associati a PD e AD in modo specifico per la malattia e il tessuto.
“I punti di rottura dei NAHR somatici che causano riarrangiamenti intracromosomici sono arricchiti in modo specifico per ogni tessuto e per ogni malattia nei geni associati a PD e AD e ciò può essere conseguente a complesse alterazioni del danno e della riparazione del DNA nella neurodegenerazione. Saranno necessari ulteriori lavori per comprendere il significato e l’impatto delle alterazioni dei profili NAHR specifici della neurodegenerazione”, spiega Piero Carninci, capo della ricerca e autore co-responsabile dello studio.
In sintesi, Pascarella e Carninci dimostrano per la prima volta che la NAHR somatica di Alu e L1 si verifica in condizioni fisiologiche in modo dipendente dal tessuto e che hotspot NAHR sono arricchiti sia nei centrosomi che negli oncogeni e nei geni soppressori del tumore. Inoltre, i risultati suggeriscono che gli eventi NAHR somatici possono innescare la differenziazione precoce dello sviluppo e la transizione da stati sani a patologici nelle cellule e nei tessuti umani. Sebbene il motivo per cui il genoma umano presenti un numero così elevato di elementi trasponibili sia ancora oggetto di dibattito, questi risultati approfondiscono la nostra comprensione delle complesse dinamiche tessuto-dipendenti dei NAHR somatici e del loro impatto sulla struttura e sulla funzione del genoma umano.