Mappiamo i punti deboli dei tumori
L’appello lanciato dagli scienziati di alcuni tra i più importanti centri di ricerca genomica del mondo per costituire la “Cancer Dependency Map”, una mappatura dei geni da cui dipende la sopravvivenza dei tumori per farne un bersaglio di farmaci. L’appello esce oggi su Nature e vede tra i suoi promotori anche Francesco Iorio, bioinformatico, group leader presso il Centro di ricerca in Biologia computazionale dello Human Technopole e team leader presso il Wellcome Sanger Institute. Human Technopole sarà il primo istituto di ricerca italiano ad aderire a questo progetto, focalizzando la ricerca sui tumori del cervello e sui metodi computazionali
Per trovare il punto debole genetico di ogni tipo di tumore è necessario analizzare un’enorme mole di dati e coinvolgere il più vasto numero di istituzioni scientifiche e ricercatori in tutto il mondo. Per questo motivo scienziati di due tra i più importanti istituti di ricerca genomica del mondo, il Wellcome Sanger Institute di Cambridge (Regno Unito) e il Broad Institute di Harvard e MIT di Cambridge, Massachusetts (USA), hanno deciso di lanciare un appello per realizzare il progetto Cancer Dependency Map: una mappatura che identifichi l’essenzialità e la funzione di geni e proteine a livello di intero genoma e in ogni tipo di tumore.
Questo lavoro avrà come obiettivo finale individuare farmaci esistenti o crearne di nuovi che combattano il cancro agendo selettivamente sui geni da cui dipendono funzioni indispensabili alla sua sopravvivenza. Per arrivare a questo risultato è stato stimato che sarà necessario “spegnere” 20.000 geni e valutare l’attività di 10.000 farmaci in almeno 20.000 modelli di cancro, misurando come queste inattivazioni cambiano la vitalità, la morfologia, l’espressione genica e altre caratteristiche delle cellule di cancro. I ricercatori stimano che la realizzazione del progetto richiederà un investimento complessivo tra i 30 e i 50 milioni di dollari in un decennio.
Tra i promotori del progetto, descritto in un articolo pubblicato oggi su Nature, c’è anche Francesco Iorio, bioinformatico, group leader presso il Centro di ricerca in Biologia computazionale dello Human Technopole e team leader presso il Wellcome Sanger Institute. Proprio lo Human Technopole sarà il primo istituto di ricerca italiano ad aderire al progetto.
Commenta Iain Mattaj, direttore dello Human Technopole: “Quasi 15 anni fa scienziati e medici hanno deciso di caratterizzare i genomi dei tumori di migliaia di pazienti attraverso progetti su larga scala come il Cancer Genome Atlas e l’International Cancer Genome Consortium. Lo sviluppo e l’applicazione della maggior parte dei farmaci antitumorali approvati recentemente hanno beneficiato dei dati generati da questi sforzi. Tuttavia, meno di un quarto delle persone con i tumori più comuni beneficia oggi della medicina di precisione. Per questa ragione abbiamo deciso di aderire al progetto Cancer Dependency Map. In Human Technopole ci focalizzeremo sui tumori cerebrali, grazie alle competenze del nostro Centro di neurogenomica guidato da Giuseppe Testa, e su metodi computazionali e di data analysis”.
Francesco Iorio, bioinformatico group leader presso il Centro di ricerca in Biologia computazionale dello Human Technopole e group leader presso il Wellcome Sanger Institute, sottolinea: “Il nostro obiettivo è audace: vogliamo valutare in laboratorio l’effetto dei farmaci e dell’intervento sui geni in ogni possibile tipo di cancro e rendere i dati accessibili ai ricercatori e agli esperti di intelligenza artificiale di tutto il mondo. Finalmente le tecnologie che oggi abbiamo a disposizione, dall’editing del genoma con la tecnologia CRISPR-Cas9 all’informatica, rendono possibile questa sfida. A condizione però di avere risorse e ricercatori a sufficienza: per questa ragione oggi lanciamo questa ‘chiamata alle armi’ ai nostri colleghi e a possibili finanziatori. Il contributo dei bioinformatici sarà molto importante: non solo perchè solo grazie ai computer saremo capaci di leggere, conservare ed elaborare l’enorme mole di informazioni derivanti da questo progetto ma anche perché dovremo fare in modo che tutti i centri coinvolti raccolgano dati secondo standard appositamente creati che li rendano comparabili tra loro e integrabili”.
Il progetto Cancer Dependency Map ha già avviato una fase pilota: sono stati analizzati decine di tipi di cancro differenti e questo lavoro ha già portato alla scoperta di più di 30 bersagli farmacologici su cui sono già stati avviati studi clinici. I dati di questa fase di sviluppo sono oggi consultati quotidianamente da circa 1.500 ricercatori in oltre 100 paesi.
I CONTENUTI DEL PROGETTO
In oncologia, una “dipendenza” è un gene, una proteina o un’altra caratteristica molecolare da cui dipende un tumore per la crescita. Potrebbe anche essere definita una vulnerabilità. Ne sono esempio geni che stimolano direttamente il riprodursi delle cellule tumorali o che regolano la produzione di proteine indispensabili per la sua crescita. Il progetto Cancer Dependency Map intende mappare sistematicamente queste vulnerabilità intervenendo direttamente su ciascun gene e proteina e misurando quello che succede nelle cellule tumorali a seguito di queste perturbazioni, in migliaia di modelli di cancro diversi, nelle diverse fasi dello sviluppo della malattia e in diversi quadri clinici. Per queste ragioni, il progetto rappresenta un passo in avanti rispetto agli studi di sequenziamento genetico dei tumori realizzati sinora perché non si limita a mappare il loro DNA ma indaga che cosa succede se si interviene su di esso.
Lo studio si avvierà espandendo, rispetto alla fase pilota, la gamma di farmaci e manipolazioni genetiche utilizzate negli esperimenti. Gli strumenti di intervento sul genoma come CRISPR possono mettere fuori uso i geni, attivarne e disattivarne l’espressione e persino manipolare le sequenze genetiche. Queste azioni potranno essere eseguite su molti tipi di tumore e contesti cellulari (come in presenza di cellule immunitarie e di altre cellule non cancerose). L’evoluzione più impegnativa del progetto sarà, in un secondo momento, l’espansione del numero di modelli di cancro studiati (linee cellulari e organoidi) rispetto al progetto pilota, che ha esaminato circa 1.000 linee di cellule tumorali.
Il progetto raccoglierà anche dati oggi insufficienti o non disponibili, come ad esempio le caratteristiche genetiche dei tipi di tumore comuni nelle popolazioni di discendenza non europea, dei tumori resistenti ai farmaci, dei tumori negli stadi iniziali della malattia, dei tumori rari o pediatrici. Saranno ricercate anche nuove strade terapeutiche: ad esempio, identificare bersagli farmacologici che promuovono la senescenza, una condizione in cui le cellule vivono ma non si riproducono più.
*************************
Nature, 28 Gennaio 2021
Cancer research needs a better map
Jesse S. Boehm (1), Mathew J. Garnett (3), David J. Adams (3), Hayley E. Francies (3), Todd R. Golub (1,2), William C. Hahn (1,2), Francesco Iorio (3,4), James M. McFarland (1) Leopold Parts (3) Francisca Vazquez (1)
- Broad Institute in Cambridge, Massachusetts, USA
- Dana- Farber Cancer Research Institute in Boston
- Wellcome Sanger Institute in Hinxton, UK.
- Human Technopole, I-20157 Milan, Italy.
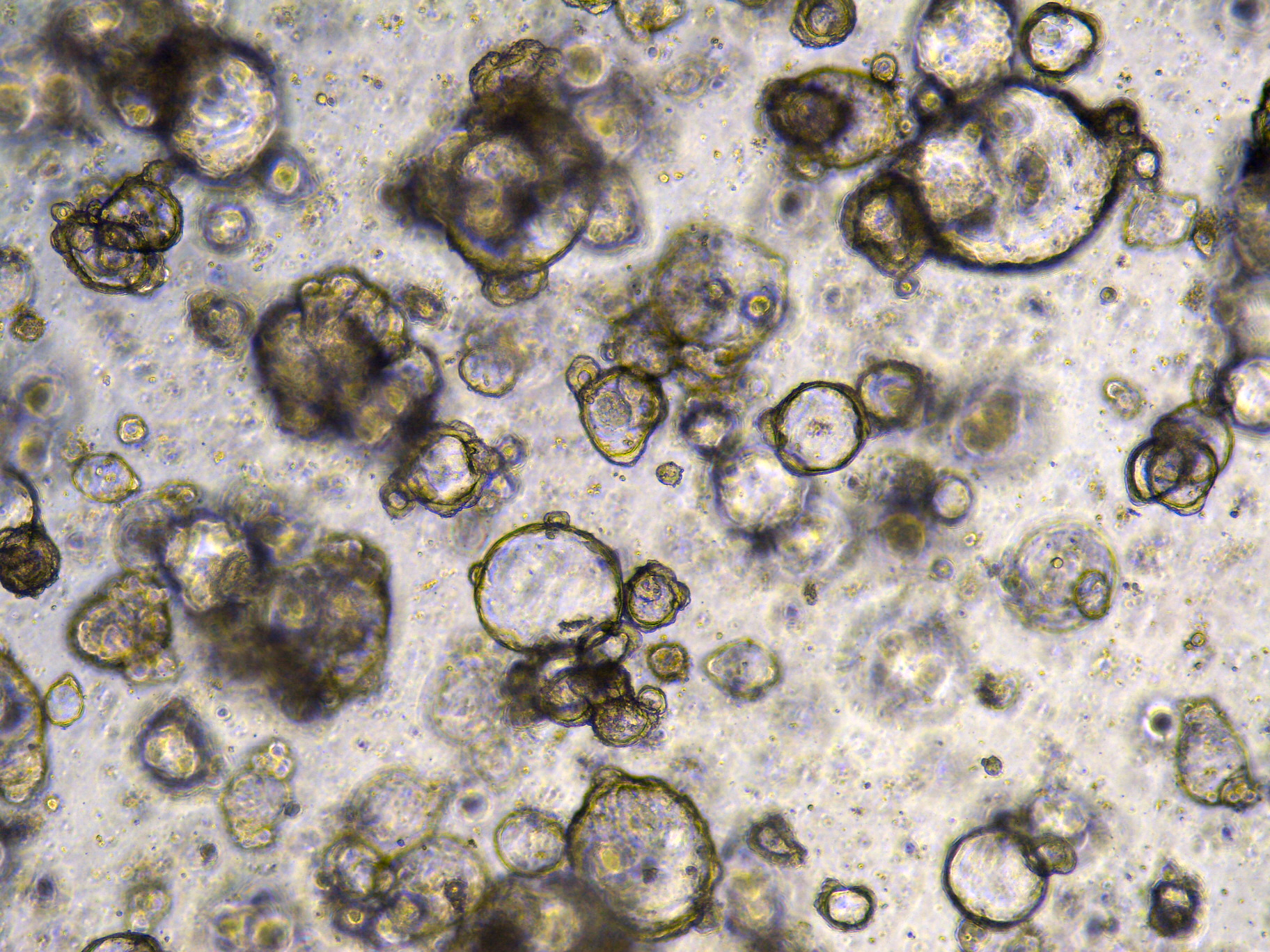
Immagine: organoidi di tumori, credit: Wellcome Sanger Institute